PACKAGES
factoextra
Cluster: PAM¶
r.pam esegue una partitioning clustering K-medoids o PAM (Partitioning Around Medoids).
PAM è un metodo più robusto rispetto a K-means, ma è più impegnativo dal punto di vista computazionale, pertanto, nel caso di matrici di dimensioni considerevoli, può essere necessario modificare i valori di default dei parametri.
Argomenti: [1]
- varlist: l’elenco delle variabili da utilizzare nell’analisi
- Determinazione del numero di cluster:
- :method => :silhouette|:wss|:gap_stat: il metodo di stima del numero ottimale di cluster (default: :gap_sta)
- :kmax => num: il numero massimo di cluster da considerare (default: 10)
- :nboot => num: il numero di Monte Carlo samples per il calcolo della statistica gap (default: 100)
- Clustering:
- :k => num|:auto: il numero di cluster da generare oppure :auto per determinare il numero dei cluster in base al metodo :silhouette (default: :auto)
- :scale => true|false: se standardizzare o meno le variabili (default: true)
- :cluster => name: il nome della variabile cluster
Tabelle disponibili:
- :analysis: riepilogo dei parametri dell’analisi
- :silh: punti con valori di silhouette negativi (associato al grafico :silh)
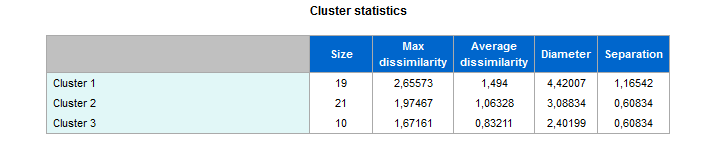
- :clus: la distribuzione dei cluster
- :med: i medoidi dei cluster
- :stats: statistiche dei cluster
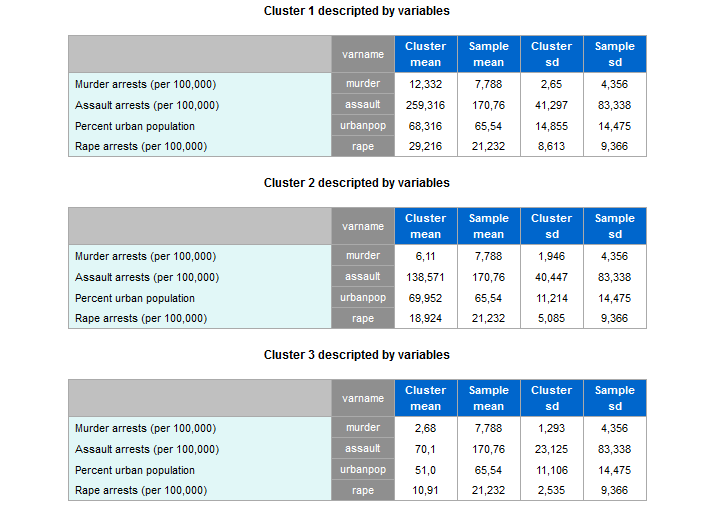
- :descrv: la descrizione dei cluster in base alle variabili originarie
Grafici disponibili:
- :dist: la matrice di dissimilarità
- :nclu: il numero ottimale di cluster
- :fctmap: i cluster plottati sul piano delle prime due componenti principali
- :silh: cluster silhouette
Metodi dell’oggetto restituito:
- centers(false|true): restituisce i centri dei cluster:
- true: come array di hash con i nomi delle variabili
- false: senza i nomi delle variabili come array di arrai (default)
1 2 | model = [:murder, :assault, :urbanpop, :rape]
r.pam model, :rownames => :state, :method => :gap_stat, :k => 3, :palette => 12
|

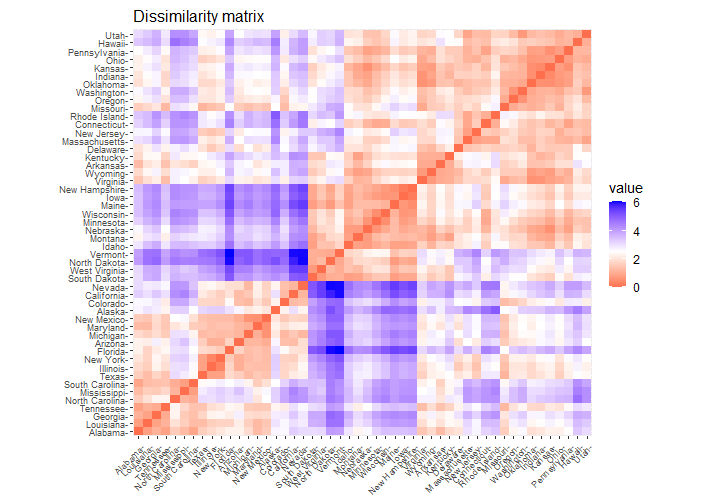

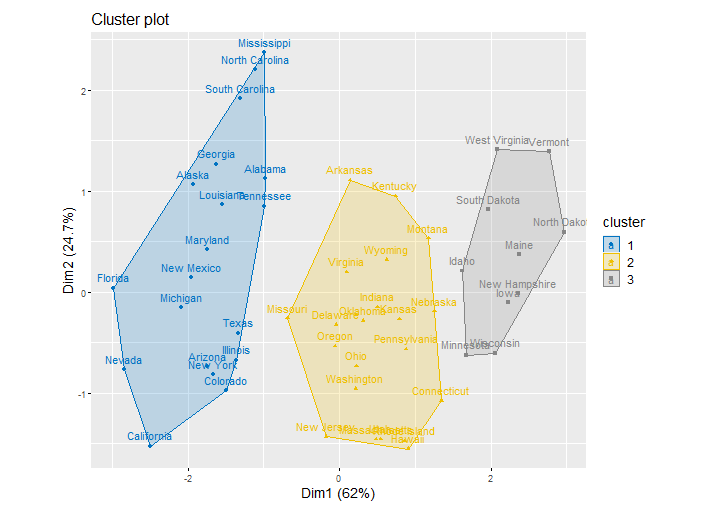
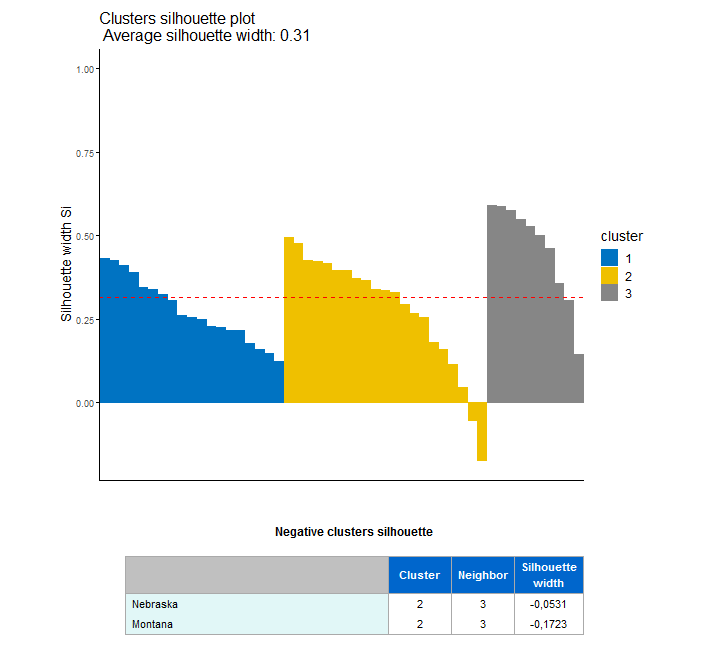

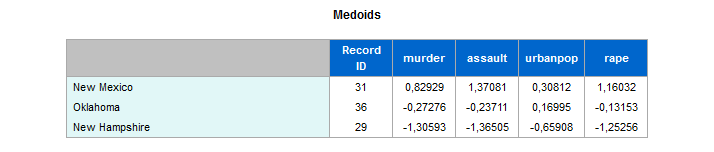
Note
| [1] | Kaufman, L. and Rousseeuw, P.J. (1990) Partitioning Around Medoids (Program PAM), in Finding Groups in Data: An Introduction to Cluster Analysis, Wiley, New York. |
| [2] | Consultare Analisi per l’elenco dei parametri generali. |